12 de Abril de 2018
Fuente: biotech-spain.com
La Esclerosis Lateral Amiotrófica (ELA) es una enfermedad neurodegenerativa que afecta a las motoneuronas implicadas en el movimiento. La pérdida de función de estas neuronas da como resultado una pérdida progresiva en la capacidad de iniciar y controlar las acciones en aquellos que la padecen, lo cual les lleva inicialmente, a no ser capaces de moverse y, posteriormente, a la muerte. Aproximadamente, uno de cada diez casos de pacientes con ELA la heredan de sus progenittores, mientras que la mayoría sufren esta enfermedad debido a la presencia de mutaciones de novo.
La identificación de los mecanismos comunes que dan como consecuencia una enfermedad es realmente esencial para desarrollar una cura. Con esto en mente, el grupo de investigación liderado por John Landers, de la UMass Medical School, y Brian Trainor, del National Institute of Aging (National Institutes of Health) ha conseguido identificar el gen KIF5A como uno de los genes implicados en el desarrollo de la ELA. Este nuevo descubrimiento permite comprender mejor la implicación de los defectos en el citoesqueleto axónico como desencadenante de esta enfermedad y abre camino al desarrollo de nuevos fármacos y terapias enfocados en las proteínas del citoesqueleto.
El equipo de investigación dirigido por Landers y Trainor llevó a cabo un estudio comparativo del genoma en más de 20.000 pacientes con ELA y aproximadamente 60.000 controles. El estudio señala el gen KIF5A como uno de los genes asociados a la ELA. Este estudio fue corroborado por el análisis de variantes raras en 1.139 casos de pacientes con ELA familiar y 19.494 individuos control.
KIF5A codifica una proteína de la familia de las quinesinas que se encarga del transporte de diferentes vesículas a través de los axones. La proteína KIF5A se sitúa unida al citoesqueleto axónico y se encarga del movimiento y anclaje de estructuras a este. Por tanto, un déficit en esta proteína impide el anclaje y transporte por el citoesqueleto del axón, lo que lleva a que las neuronas no sean capaces de comunicarse correctamente con las células post-sinápticas. Además, la proteína KIF5A también se encarga del transporte de proteínas que se unen a ADN y ARN, convirtiéndose este hecho en otra posible causa de la aparición de la ELA.
Defectos similares del citoesqueleto producidos por mutaciones en otros genes diferentes al KIF5A han sido descritos en otros estudios como causantes de la ELA. Como explica Landers, “Las mutaciones en KIF5A se unen a una lista en crecimiento de mutaciones genéticas relacionadas conocidas y refuerza la evidencia del papel que tienen los defectos en el citoesqueleto en la patogénesis de la ELA”. Esta enfermedad, además de poder ser causada por defectos en el citoesqueleto, también puede ser producida por alteraciones en otras dos rutas biológicas en las cuales ya se conocen algunos de los genes implicados. Estas dos rutas adicionales serían el metabolismo del ARN (genes C9orf72, TDP-43, FUS, HNRNPA1, y MATR) y la homeostasis proteica (genes UBQLN2, VCP, OPTN, y VAPB).
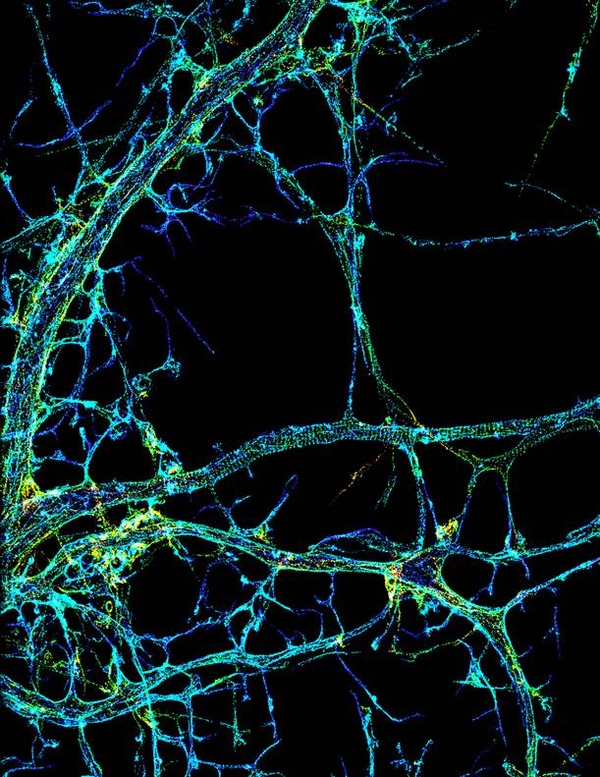
El descubrimiento de un nuevo gen causante de ELA permite relacionar esta enfermedad con el citoesqueleto. Imagen: citoesqueleto de axones de células nerviosasXiaowei Zhuang Laboratory, Howard Hughes Medical Institute, Harvard University.
Lo curioso de este descubrimiento es que ya se habían descrito mutaciones en el gen KIF5A como causa de otras enfermedades, la paraplejia espástica hereditaria (HSP) y la enfermedad de Charcot-Marie tipo 2 (CMT2). En el caso de HPS y CMT2, las mutaciones se encuentran en la región del gen que codifica para el extremo N-terminal, mientras que en la ELA, las mutaciones desembocan en proteínas alteradas en el extremo C-terminal. Así, estas tres enfermedades difieren en que HPS y CMT2 son causadas por mutaciones que modifican la base de la proteína KIF5A (la región que se une al citoesqueleto) y la ELA por mutaciones en el extremo de unión a las moléculas que transporta la proteína. La mutación descrita en el artículo del grupo de investigación de Landers y Trainor no es tan grave como otras mutaciones en otros genes, ya que los pacientes que la padecen viven de media unos 10 años tras el diagnóstico, en contraste con los 2-4 años para otras mutaciones.
El próximo paso para Landers y Trainor es investigar y conocer mejor por qué una mutación en KIF5A que causa una proteína alterada en su extremo C-Terminal da lugar a pacientes con ELA, mientras que mutaciones que modifican un único aminoácido en la base de la proteína causan HSP. Con este objetivo en mente y el fin de elaborar fármacos contra estas enfermedades, los investigadores han comenzado a desarrollar modelos animales con las mutaciones en KIF5A que dan lugar tanto a HSP y CMT2 como a ELA. Además, en paralelo, comenzarán a realizar estudios comparativos en los que se utilizarán células madre de pacientes con ELA y pacientes con HSP y CMT2.